近日,欧宝app体育官网入口ios
生命科学与健康工程学院潘军华教授团队在《Cell》在线发表了题为“Viral DNA polymerase structures reveal mechanisms of antiviral drug resistance”(病毒DNA聚合酶结构揭示抗病毒药物耐药机制)的研究论文(原文链接:https://www.cell.com/cell/fulltext/S0092-8674(24)00842-0),为我校首次在三大顶刊之一的《Cell》上发表研究成果。潘军华教授为共同第一作者,哈佛医学院为第一通讯单位,欧宝app体育官网入口ios
为第二通讯单位。

该研究利用冷冻电镜(cryo-EM)技术发现了疱疹病毒聚合酶全酶复合物多种构象并存的现象(图1),并通过解析一系列高分辨率结构及详细的结构生物学研究与结构-功能分析阐明了DNA聚合酶的结构动态影响药物选择性及抗药性的分子机理以及该酶高效延续合成DNA的结构生物学原理。
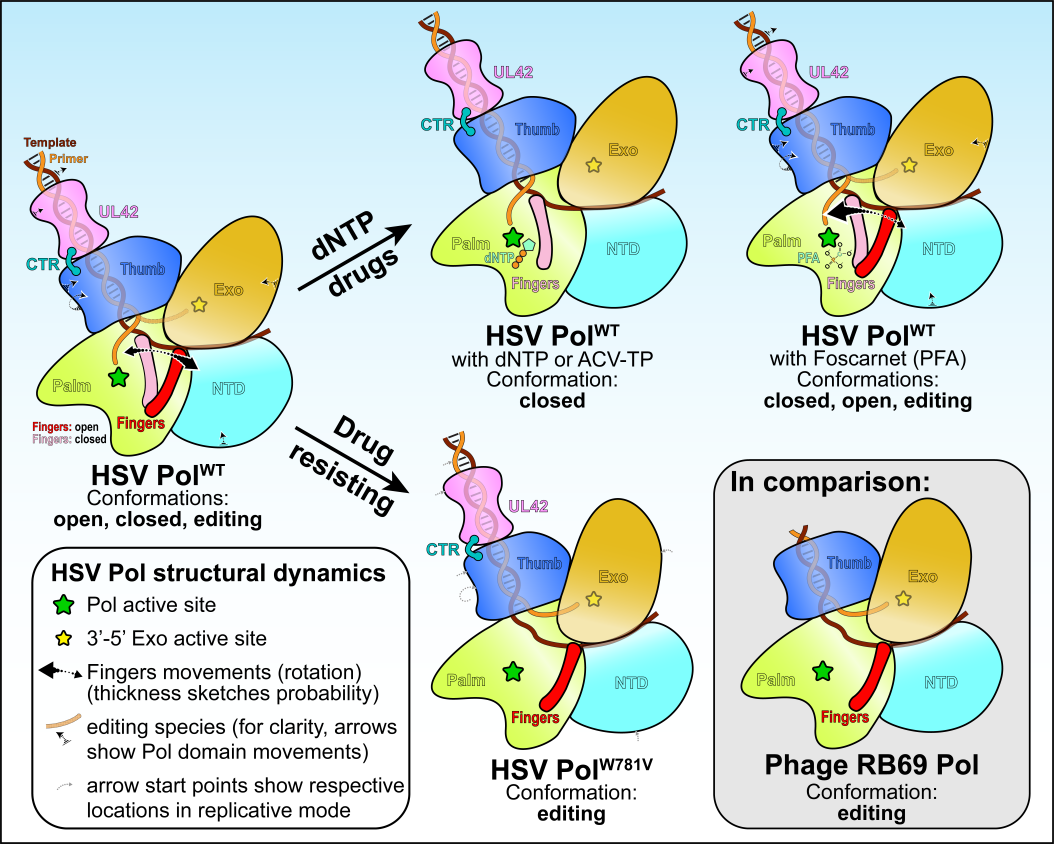
图1疱疹病毒聚合酶全酶复合物的多种构象
疱疹病毒的聚合酶是治疗该病毒的主要药物阿昔洛韦(Acyclovir)的三磷酸形式以及二线药物膦甲酸钠(Foscarnet)的靶标。疱疹病毒聚合酶(UL30或Pol)催化亚基多个位点的碱基替换均能导致临床上对这两种药物的抗性。但这些突变如何导致药物抗性以及阿昔洛韦和膦甲酸钠如何选择性地对疱疹病毒的聚合酶产生响应尚不明确。针对上述问题,本研究利用cryo-EM技术解析了结合DNA的疱疹病毒聚合酶全酶复合物的高分辨率结构(图2),并揭示了该复合物的多种不同构象。这些结构阐明了催化亚基Pol和持续合成因子UL42如何结合DNA以促进DNA持续合成的机理。本研究还发现在没有引入核苷酸的情况下Pol在多种构象中呈现指状结构域的闭合状态。药物结合结构揭示了抗病毒药物如何选择性地结合更易采用闭合构象的酶。分子动力学模拟和耐药突变体的cryo-EM结构表明,一些耐药突变体通过调节构象动力学影响药物选择性,这些研究阐明了疱疹病毒DNA聚合酶驱动药物选择性的机制。
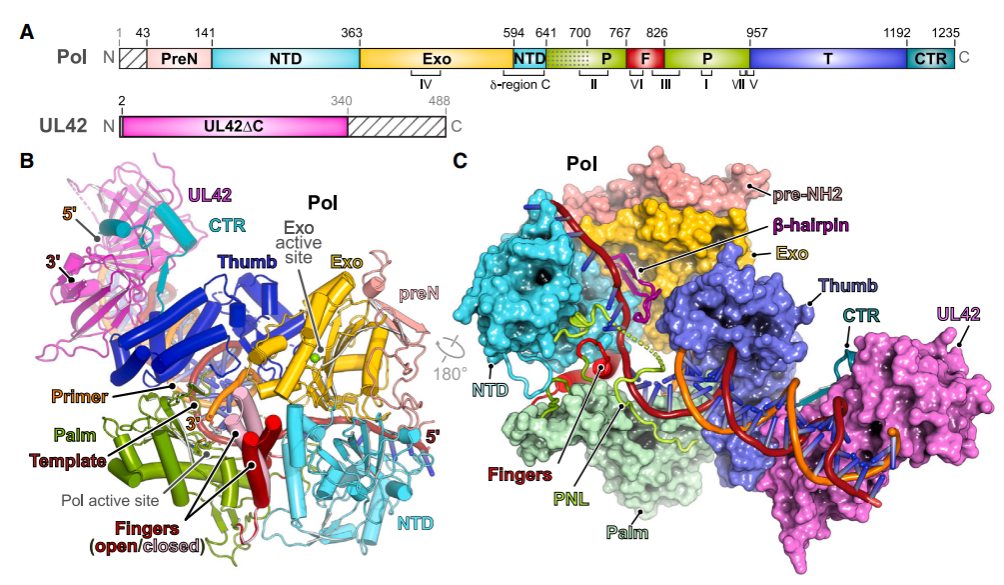
图2结合DNA的疱疹病毒聚合酶全酶的结构
本研究报道了在引入或缺失核苷酸的情况下疱疹病毒Pol-UL42异源二聚体结合DNA引物-模板的高分辨率结构。这些结构的解析有助于阐明该病毒聚合酶持续进行DNA合成的机制,并可作为模板帮助设计捕获Pol不同构象状态的变构抑制剂,从而对抗抗病毒药物产生的新的耐药性。
潘军华教授为我校2023年从海外重点引进的高层次人才,是结构生物学、生物化学和病毒学专家。潘教授以第一作者或通讯作者在Nature、Science、Cell、PNAS、Structure、Biophysics J、J Struct Biol、J Virol和Virology等杂志发表论文十余篇,拥有专利一项,担任Nature Communications、Communications Biology和Bioconjugate Chemistry等杂志审稿人。
责任编辑:陈凌
